El Hospital Niño Jesús creará ‘gemelos virtuales’ de niños con leucemia para simular su respuesta al tratamiento
El Hospital Infantil Universitario Niño Jesús coordina un proyecto de investigación, llamado ‘Leukodomics’, que tiene como objetivo crear ‘gemelos virtuales’ de niños y adolescentes con leucemia para poder simular su respuesta a cada tratamiento y predecir sus probabilidades de éxito, posibles toxicidades o su evolución a largo plazo.
Para ello, se generarán unos modelos digitales que integrarán la información del paciente (cuál fue el diagnóstico, cómo fue la respuesta al tratamiento o cuál es su fondo genético) y la de sus células malignas (mutaciones genéticas, subprogramas de expresión génica, arquitectura del genoma y características biomecánicas).
Se trata de un modelo personalizado de predicción de evolución de la enfermedad en diferentes escenarios simulados, sin necesidad de la intervención directa del paciente, minimizando el nivel de riesgo terapéutico.
Será la primera vez que se incorporan a la investigación de la leucemia infantil modelos computacionales de enfermedad y recursos informáticos procedentes de la ciencia de datos. El objetivo final es aprovechar los nuevos recursos informáticos y tecnológicos para mejorar los resultados de los tratamientos.
Una vez que se desarrollen estos ‘gemelos virtuales’, en principio con el estudio de aproximadamente 100 pacientes, se deberá realizar un ensayo clínico para comprobar, con evidencia científica, cuál es el grado de efectividad de la herramienta para prever la evolución del paciente. Así, si el proyecto es exitoso, se podría validar para poder usarla en la consulta del oncólogo “en unos 4 o 5 años”.
En rueda de prensa, Manuel Ramírez Orellana, coordinador de la investigación y jefe de la Sección de Oncología y director de Terapias Avanzadas del Hospital Niño Jesús, ha explicado que el proyecto pretende integrar “toda la información que existe alrededor de los niños y adolescentes con leucemia, no solo su enfermedad, sino la biología de sus células, su constitución genética, el tratamiento, la respuesta que ha tenido y aspectos nuevos, como características poco consideradas anteriormente”.
Con toda esta información, se generaría ese modelo individual de cada paciente. “Es como si pudiéramos tener un gemelo de cada niño o adolescente con leucemia”, ha detallado el investigador.
De esta forma, se ayudaría al oncólogo pediátrico “en el día a día del trato con estos pacientes, de manera que puedan simular en un ordenador o una aplicación situaciones que se dan en la toma de decisiones”. “Pensamos que interesaría mucho poder hacer estos experimentos antes de tomar la decisión final en un niño”, ha resaltado.
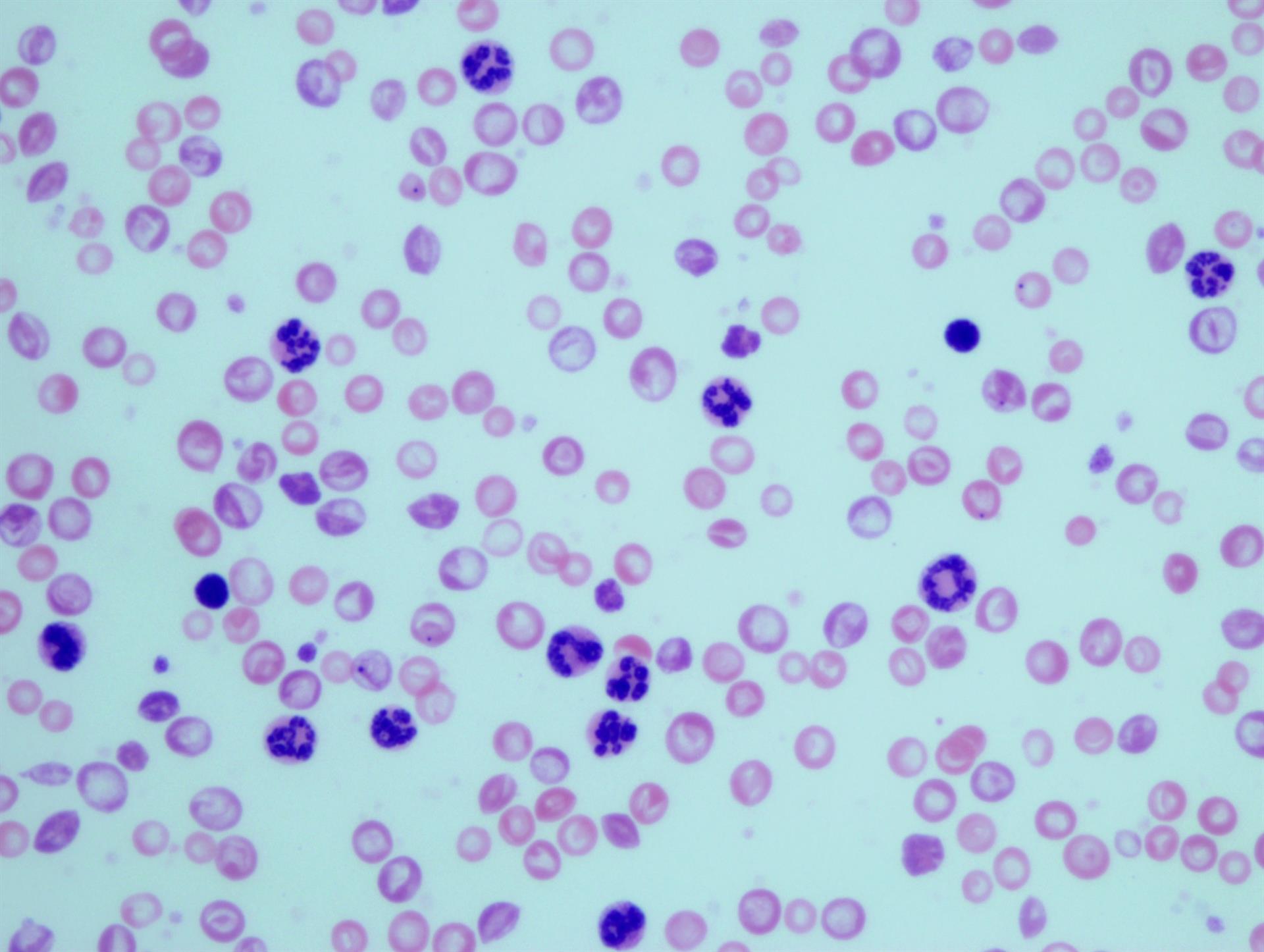
Francisco Monroy, catedrático de la Universidad Complutense de Madrid (UCM) y director de Biofísica Traslacional del Instituto de Investigación Hospital 12 de Octubre i+12, ha recordado que la leucemia es una enfermedad “grave y maligna” que nace en la médula ósea de los pacientes y que, de alguna manera, altera los rasgos de los glóbulos blancos (leucocitos).
Tal y como ha detallado, en determinados casos, pese a haber un tratamiento exitoso, normalmente realizado con quimioterapia, los pacientes pueden experimentar una recaída a los pocos años del tratamiento y aparecer complicaciones como la metástasis.
“Esas inesperadas consecuencias vienen motivadas por la existencia de unos rasgos que somos capaces de identificar de forma analógica, mirándolas en el laboratorio”, ha esgrimido, a lo que ha puntualizado que ahora, gracias a este proyecto, quieren aprovechar la digitalización para gestionar toda esta información desde el ordenador.
En este sentido, Carlos Torroja Fungairiño, investigador del Centro Nacional de Investigación Cardiovascular (CNIC), ha señalado que las nuevas técnicas de investigación, que ya están disponibles y se podrán utilizar en el proyecto, “son capaces de analizar miles y miles de células desde el punto de vista genético”. “Lo único que podíamos hacer hasta entonces era analizar un conjunto de células todas juntas. Ahora podemos ver cada una individualmente”, ha agregado.
El investigador ha continuado sosteniendo que el objetivo de la nueva investigación es “incorporar la información genética, entre otras cosas, para entender qué pasa cuando un paciente no reacciona a la terapia. Nunca se sabe, dadas las condiciones y los parámetros que se conocen actualmente, por qué no”, ha afirmado.
Diego Herráez Aguilar, profesor en la Universidad Francisco de Vitoria e investigador principal del Grupo de Biofísica Computacional (BioCompLab) en Pozuelo de Alarcón, ha añadido que, además de la información genética, también se incluirá la biofísica, es decir, el “sustrato físico de las células que tienen una patología”.
Su papel dentro del proyecto de investigación será desarrollar nuevas técnicas para “coger las fuentes de datos biofísicos y hacer una traducción”. “Algo que cada día tenemos más claro es que los datos son oro. Quien posee los datos tiene un avance grandísimo en cualquier proceso de análisis y toma de decisiones”, ha remachado.
El Hospital Niño Jesús ha reunido a especialistas en Informática, Matemáticas, Física y Biología. Se trata de un consorcio integrado por expertos de la Universidad Complutense de Madrid y el 1+12, la Universidad de Castilla-La Mancha, la Universidad Francisco de Vitoria y el CNIC, coordinados por oncólogos pediátricos del Niño Jesús.
Este proyecto, financiado a través de fondos europeos, se inició el pasado mes de diciembre y tendrá una duración de dos años de investigación aplicada e intensiva en leucemia infantil.